ЯÉCIPROCS, le groupe thématique de biologie de l'AFC et le Cambridge Crystallographic Data Centre (CCDC) ont organisé avec le soutien de la MITI du CNRS, de l'AFC et de l'UFR des sciences de l'Université de Versailles Saint Quentin (UVSQ) une formation aux outils de la base structurale de Cambridge, CSD. Cette réunion s'est tenue le 23 aoüt 2022 sur le très beau campus de l'UVSQ à Versailles grâce au soutien très efficace du comité local d'organisation constitué de Nathalie Guillou et Clément Falaise.
C'est dans une ambiance studieuse et décontractée que 34 participants (il y avait eu 42 préinscriptions) et 4 intervenants du CCDC ont interagi au cours d'une journée dense et intense. L'accessibilité et la disponibilité des formateurs ont été les atouts majeurs de cette formation. Il faut noter que pour ЯÉCIPROCS c'était la première fois que les aspects de la biologie structurale étaient abordés et cela était bien évidemment lié à l'augmentation de périmètre du réseau suite à son intégration en année d'incubation à la MITI.
Suzanna Ward a commencé par présenter la Base Structurale de Cambridge (CSD) et son positionnement vis à vis des 3 autres grandes bases de données : PDB, ICSD et ICDD. Elle a poursuivi par la présentation des logiciels Mercury et Hermes qui sont conçus respectivement pour les structures de petites et de macro molécules. Elle a ensuite présenté dans les grandes lignes les fonctionnalités de Mercury. Ilaria Gimondi a ensuite pris le relai pour entrer plus avant dans les fonctionnalités de Mercury.
Sur le même modèle d'intervention Suzanna Ward et Ilaria Gimondi ont présenté Hermes et ses fonctionnalités. Un premier TP co-encadré par Natalie Johnson et Andy Maloney a suivi sur l'utilisation de Mercury avant que Suzanna Ward aborde la visualisation des liaisons Hydrogène dans Mercury. Un deuxième TP sur Mercury permettait d'atteindre une pause-café bien méritée.
La fin de la matinée a permis à Suzanna Ward de présenter le module Mogul et ses capacités d'analyses de configurations moléculaires en se servant de la CSD et aux participants de l'utiliser au cours d'un TP. La session s’est terminée dans une joyeuse atmosphère par une évaluation quizz interactive et le lancement d'un concours de 15 minutes uniquement pour la meilleure représentation moléculaire avec Mercury. La gagnante – choisie par les participants – a été Asma Mansouri de l’Institut des Matériaux Poreux de Paris (IMAP UMR 8004, Ecole Normale Supérieure, ESPCI Paris, CNRS, PSL University). Son prix : eternal glory !
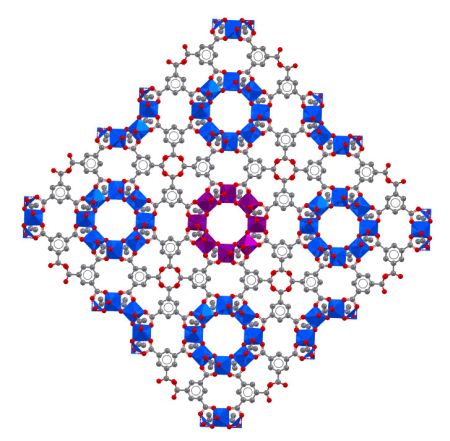
Représentation structurale d'un MOF par Asma Mansouri (reprise avec permission)
Après un magnifique buffet froid et quelques problèmes de projecteur la session de l'après-midi a débuté par Suzanna Ward montrant comment explorer la structure comme par exemple dans le cas de polymorphismes. Ilaria Gimondi a approfondi le sujet en présentant les Full Interaction Maps (FIMS). Après une courte pause Suzanna Ward a abordé les interactions protéines ligands via le module Gold. Un dernier TP a suivi au choix soit sur les FIMS soit sur les interactions protéines ligands avec Gold. La session s’est terminée, toujours dans une très bonne atmosphère, par un dernier quizz et le remplissage d'un formulaire d'évaluation de la formation.
En conclusion cette formation de très haut niveau de par la qualité des intervenants et de la qualité pédagogique des supports a su capter l'auditoire qui a découvert de nombreuses fonctionnalités insoupçonnées des logiciels du CCDC, et le transfert de connaissance via les TP semble acquis et durable.
Christian Philouze, DCM, Université de Grenoble





