Auteurs: Bruno Landeros-Rivera, Julia Contreras-García
Laboratoire de Chimie Théorique, Paris, France
Téléchargement du logiciel NCIPLOT
I. Introduction et principe du calcul.
NCI [1,2] est un descripteur basé sur la densité électronique ρ permettant la visualisation des interactions non-covalentes. Les interactions non-covalentes sont identifiées par des valeurs réduites de ρ et de s = |∇ρ|/(cρ4/3) qui engendrent des piques dans un graph s(ρ) [Figure 1 en bas]. La visualisation en 3D des points associés aux valeurs évoquées précédemment, donne lieu à des isosurfaces, qui révèlent les interactions non-covalentes [Figure 1 en haut]. Ainsi, NCI facilite l’identification des liaisons hydrogènes, des -stacking [illustrés en Figure 1] mais également des liaisons halogène, des cation-π, etc.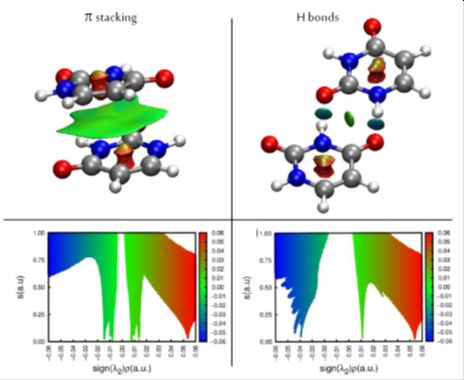
Figure 1. NCI pour le dimère d’uracile en forme π-stacking (gauche) et avec des liaisons hydrogène (droite). En haut, des isosurfaces de s revèlent les interactions faibles. En bas, les diagrammes permettent d’identifier les piques où s devient nul.
Il existe plusieurs variantes suivant le type de données que nous voulons traiter :
|
Programme |
Données |
Format d’input |
Références |
|
NCIPLOT |
-géométrie (approximation IAM) |
xyz |
|
|
-fonction d’onde moléculaire |
wfn,wfx |
||
 |
-données macromoléculaires |
pdb |
[5] |
Dans tous les cas, une grille est construite autour de la molécule et les valeurs de et sont calculées sur chaque point de la grille. Puisque seuls les points de basse densité et bas gradient réduit sont d’intérêt, des grilles adaptatives ont été implémentés afin d’analyser uniquement les régions intéressantes du point de vue des interactions non-covalentes, évitant ainsi de calculer un grand nombre de points inintéressants. Ces grilles adaptatives réduisent énormément le coût du calcul, donnant la possibilité d’étudier des systèmes de grande taille.
Installation
Pour installer NCIPLOT-4.0 [4] sous un environnement Linux (natif ou sur la couche Linux WSL intégrée à WIndows-10) une fois dans le répertoire d’installation :
- modifier Makefile.inc pour choisir votre compilateur Fortran
- make
Alternativement, un exécutable statique nciplot.x est aussi distribué dans le fichier de téléchargement.
Execution
L’input basique pour NCIPLOT-4.0 est composé de deux parties: la première contenant le nombre de fichiers à lire et la deuxième les noms des fichiers. Les fichiers .xyz se créent facilement à partir d'un fichier .cif, p.e. Par exemple :
2Benzene1.xyz
Benzene2.xyz
Le code reconnaît l’extension du fichier et reconstruit la densité IAM (c'est-à-dire la densité promoléculaire) à partir de coordonnées géométriques. Pour l’exécuter il suffit d’écrire :
nciplot.x < input.file [> output.file]
On obtient 4 fichiers : un fichier .dat avec les données pour créer des graph comme celui en Figure 1 en haut, deux fichiers .cube pour la visualisation en 3D comme illustrés en Figure 1 en bas et 1 fichier .vmd pour la lecture des fichiers directe avec le programme de visualisation moléculaire VMD.
Cet input permet de visualiser toutes les interactions non-covalentes (intra et intermoléculaires). Ceci est illustré en Figure 2 pour le fluconazole (FLC), un fongicide. Deux molécules ont été extraites du système cristallin (polymorphe 7, Ref [6]) et leurs interactions non-covalentes sont représentées dans la Figure 2a. Les isosurfaces étendues de couleur verte correspondent à des interactions van der Waals tandis que celles de couleur bleue sont associées aux liaisons hydrogènes. Les répulsions stériques apparaissent en rouge.
Afin de peaufiner le type d’interaction, plusieurs options sont disponibles. Le mot clé INTERMOLECULAR permet de visualiser uniquement les interactions intermoléculaires. Ainsi, dans la Figure 2b seules les répulsions stériques sont visualisées. Les mots clé CUBE et RADIUS permettent de choisir une partie de l’espace spécifique. Dans la Figure 2c on opte pour un cube centré au milieu de la liaison hydrogène N-H, avec un rayon de 1 Å afin de nous focaliser sur cette liaison. Finalement, la visualisation peut également se restreindre aux interactions d’une certaine force (un intervalle de densités) avec le mot clé CUTPLOT. Dans la Figure 2d nous avons choisi de nous focaliser sur les interactions de van der Waals du dimère, omettant toutes les interactions avec une densité inferieure à r=0.025 a.u.
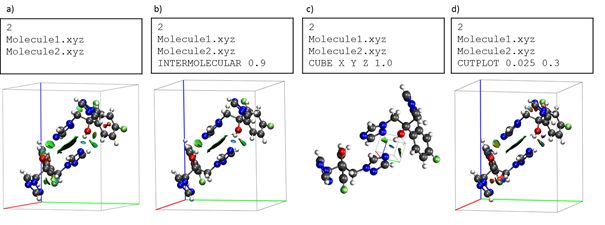
Figure 2. Options de mots clés décrites dans le texte et les différents résultats qu’elles produisent.
L’étude d’ancrage d’une petite molécule, comme le fluconazole, dans son récepteur peut être mené facilement à l’aide du mot clé LIGAND. Dans la Figure 3 on représente les interactions du fluconazole lié à l’enzyme Saccharomyces cerevisiae Lanosterol 14α-Demethylase (CYP51) [PDB extrait de la Ref. [7]). Deux mots-clés (CG2FG et INCREMENTS) permettent d’accélérer le calcul pour un système de grande taille. Le mot clé INCREMENTS réduit la résolution tandis que le mot clé CG2FG permet l’utilisation de 4 grilles adaptatives..
Dans la figure 3, l’enzyme se lie au site active de la protéine à travers des interactions non covalentes spécifiques : une liaison dative entre le l’azote d’une groupe imidazole du FLC et l’atome de fer du cofacteur hème du CYP51 ainsi que des liaisons hydrogène avec des molécules d’eaux présentes dans le site active. Ces interactions ont été mises en valeur avec des carrés bleus dans l’input de la Figure 3. Il y a aussi un grand nombre d’interactions de van der Waals qui soulignent la forme complémentaire du médicament avec le site active. Puisque cette protéine est impliquée dans la synthèse du ergosterol, nécessaire pour un bon fonctionnement des membranes du champignon, l’ensemble de ces interactions permet au fongicide d’accomplir sa tâche thérapeutique ralentissant sa croissance.
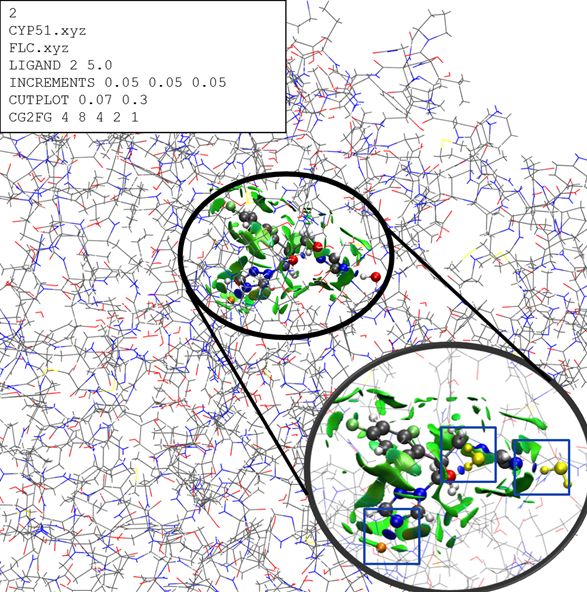
Figure 3. NCI du fluconazol lié au site active de l’enzyme Saccharomyces cerevisiae Lanosterol 14α-Demethylase (CYP51). L’encadré supérieur illustre les mots clés utilisés pour produire la figure. L’encadré inférieur équivaut à un élargissement des interactions, avec les interactions les plus fortes mises en valeur par des carreaux bleus (molécules d’eaux en jaune et atome de fer en orange).
Le volume et la densité associés aux surfaces NCI peuvent être obtenus par simple intégration des régions impliquées. L’intégration d’une région spécifique est invoqué avec le mot clé RANGE. Si un chiffre supérieur à 1 est utilisé, des intervalles de densité sont définis dans l’intégration afin de séparer les volumes en fonction de la force de la liaison. Par exemple, pour obtenir les volumes et les charges des interactions non-covalentes associées aux liaisons fortes, à des interactions de van der Waals et aux interactions stériques, il suffit de rajouter à l’input de la Figure 3 quelques lignes :
2
CYP51.xyz
FLC.xyz
LIGAND 2 5.0
INCREMENTS 0.05 0.05 0.05
CUTPLOT 0.07 0.3
CG2FG 4 8 4 2 1
RANGE 3
-0.1 -0.02
-0.02 0.02
0.02 0.1
Avec cette input, on obtient un intégrale de ρ(n=1) avec trois intervalles: de sign(λ2)ρ=-0.1 à -0.02 a.u., -0.02 à 0.02 et 0.02 à 0.1. Le signe négatif de la valeur propre λ2 de la matrice hessienne de ρ indique des interactions attractives, tandis que le signe positif correspond aux interactions répulsives. Les charges suivantes des interactions fortes (liaison hydrogènes, datives), van der Waals et stériques sont respectivement égales à 1.6112, 5.9103 and 0.7856 a.u. Les charges et la force des interactions confirment les observations faites à partir de la Figure 3: les interactions van der Waals qui déterminent la complémentarité du FLC au site active du CYP51 jouent un rôle prépondérant dans l’activité du fongicide.
III. NCIweb
Afin de faciliter l’utilisation de NCI sur des systèmes étendues, nous avons développé une interface graphique, NCIweb, qui permet de télécharger directement les pdb, le nettoyer, et choisir les interactions d’intérêt [Figure 4].
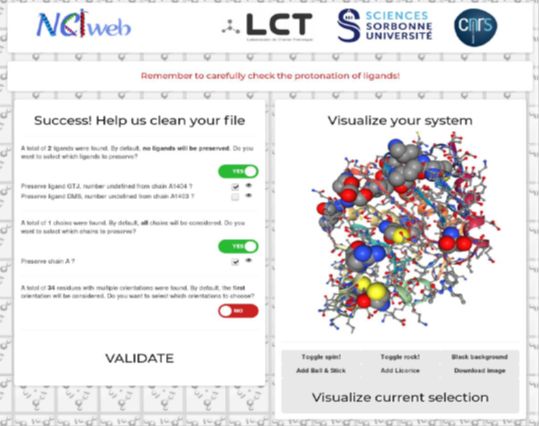
Figure 4. Interface graphique de NCIweb
III. Disponibilité
Références
[1] E. R. Johnson, S. Keinan, P. Mori-Sanchez, J. Contreras-Garcia, A. J. Cohen, and W. Yang, “Revealing Noncovalent Interactions”, J. Am. Chem. Soc. 132 , 6498 (2010).
[2] R. Laplaza, F. Peccati, R. A. Boto, C. Quan, A. Carbone, J.-P. Piquemal, Y. Maday, J. Contreras-García “NCIPLOT and the analysis of Noncovalent Interactions using the Reduced Density Gradient”, WIREs (submitted)
[3] J. Contreras-Garcia, E. Johnson, S. Keinan, R. Chaudret, J-P Piquemal, D. Beratan, W. Yang, “NCIPLOT: a program for plotting non-covalent interaction regions”, J. Chem. Theor. Comp. 7, 625 (2011)
[4] R. A. Boto, F. Peccati, R. Laplaza, C. Quan, A. Carbone, J.-P. Piquemal, Y. Maday, J. Contreras-Garcia*, “NCIPLOT4: A new step towards a fast quantification of noncovalent interactions” (sous presse)
[5] https://www.lct.jussieu.fr/pagesperso/contrera/nciweb/index.php
[6] M. Karanam, S. Dev, and A. R. Choudhury, “New Polymorphs of Fluconazole: Results from Cocrystallization Experiments”, Cryst. Growth Des. 12, 240 (2012).
[7] A. A. Sagatova, M. V. Keniya, R. K. Wilson, B. C. Monk, and J. D. A. Tyndall, “Structural Insights into Binding of the Antifungal Drug Fluconazole to Saccharomyces cerevisiae Lanosterol 14α-Demethylase”, Antimicrob. Agents Chemother. 59, 4982 (2015).





