Le virus, SARS-COV-2, responsable de la pandémie de Covid-19 qui progresse un peu plus chaque jour dans le monde, appartient à la famille des coronavirus dont les premiers membres ont été identifiés il y a près d’un siècle [1]. Ces virus possédant un génome à ARN (acide ribonucléique), sont entourés d’une enveloppe membranaire et portent à leur surface des protubérances, reconnaissables en microscopie électronique et à l’origine de leur appellation (car donnant un aspect de couronne ; Fig. 1). Ces protubérances correspondent à la glycoprotéine de surface S (pour Spike) qui joue un rôle clé dans l’entrée du virus dans les cellules hôtes.
Depuis quelques semaines, partout dans le monde, la communauté médicale et scientifique est fortement mobilisée afin de mieux comprendre les spécificités de ce nouveau virus, SARS-COV-2, et trouver de nouvelles stratégies thérapeutiques et préventives pour mettre un terme à cette pandémie. Parmi ces chercheurs, les bio-structuralistes jouent un rôle essentiel en déterminant les structures à l’échelle atomique des principales protéines de SARS-COV-2 par diffraction de rayons X et/ou par cryo-microscopie électronique (cryo-ME). Les deux principales protéines faisant l’objet d’études structurales sont la glycoprotéine S et une protéase (enzyme capable de couper d’autres protéines) essentielle du virus, permettant la production de plusieurs protéines virales à partir d’une seule polyprotéine.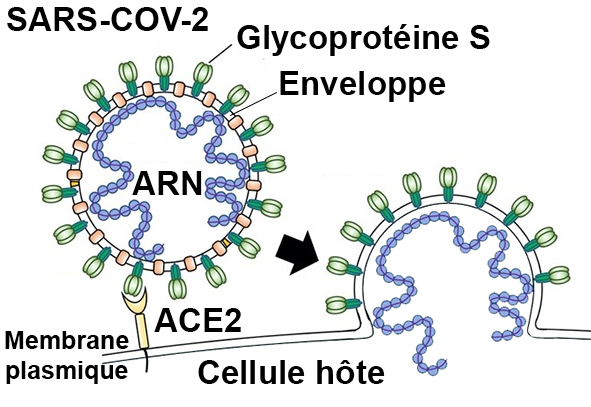
Se trouvant à la surface du virus, la glycoprotéine S permet l’entrée du virus dans les cellules humaines via son interaction avec un récepteur, identifié comme étant l’enzyme ACE2, présent à la surface des cellules infectées [2]. Au cours du mois de mars 2020, plusieurs structures partielles de la glycoprotéine S, seule ou en complexe avec son récepteur ACE2, ont été déterminées par cryo-ME révélant ainsi son mode de reconnaissance du récepteur et les mécanismes moléculaires permettant la fusion de l’enveloppe virale avec la membrane plasmique des cellules infectées [2,3,4]. Ces informations structurales pourront maintenant être exploitées pour d’une part l’identification d’inhibiteurs de ces étapes clés du cycle viral, qui à terme pourraient être utilisés comme antiviraux, et d’autre part, pour le développement de vaccins. Étant à la surface de ces virus, la glycoprotéine S joue un rôle essentiel dans la réponse immunitaire de l’hôte. Les principaux anticorps neutralisants reconnaissent en effet cette protéine. Le développement d’un vaccin efficace contre SARS-COV-2 nécessitera d’identifier les régions de la glycoprotéine S ayant un fort potentiel immunogène et entrainant une réponse immunitaire efficace et spécifique contre ce virus.
Une deuxième protéine qui fait l’objet de nombreuses études structurales est la principale protéase de SARS-COV-2, appelée MPRO ou 3CLPRO, qui représente actuellement une des cibles thérapeutiques les plus prometteuses pour le développement d’antiviraux. Dans un premier temps, la structure cristalline de cette enzyme a été déterminée à haute résolution [5]. Désormais, de nombreuses équipes de recherche visent à identifier des inhibiteurs spécifiques et efficaces de cette protéase par des approches théoriques (modélisation, in silico drug design etc) [6] et/ou expérimentales, notamment par la détermination de structures cristallines de complexes entre la protéase et des petites molécules inhibitrices. Dans ce contexte, de grandes quantités de données cristallographiques ont été enregistrées par l’équipe XChem du synchrotron Diamond en Grande-Bretagne suite à un criblage haut-débit de petites molécules, et un appel aux volontaires a été lancé aux cristallographes, bio-structuralistes et chimistes qui souhaiteraient prendre part à l’analyse de ces données, au design de nouvelles molécules ou à la synthèse d’inhibiteurs.
Alors que les grands instruments nationaux sont fermés depuis le début du confinement, ils restent néanmoins mobilisés eux aussi pour lutter contre le Covid-19. Le synchrotron SOLEIL, par exemple, a mis en place un mécanisme de réouverture pour de courtes périodes pour des projets en lien avec le Covid-19 via le système de proposition d’Accès Rapide.
Pour tous les chercheurs souhaitant mettre à profit leurs expertises pour combattre le Covid-19, vous pouvez vous rendre sur le site : https://crowdfightcovid19.org/. Les chercheurs du CRCA – CBI à Toulouse ont mis en place cette plateforme ayant pour objectif de mettre les compétences des chercheurs non mobilisés sur le Covid-19 au service de ceux déjà engagés dans cette lutte.
Texte: Joanna Timmins (Institut de Biologie Structurale, Virus Infection & Cancer Group, DNA Damage & Repair Team, Grenoble)
2 avril 2020
Références
- Gozlan M. Il était une fois les coronavirus. Blog du Monde. 2020 Mar 27.
- Walls AC, Park YJ, Tortorici MA, Wall A, McGuire AT, Veesler D. Structure, Function, and Antigenicity of the SARS-CoV-2 Spike Glycoprotein. Cell. 2020 Mar 6. pii: S0092-8674(20)30262-2.
- Yan R, Zhang Y, Li Y, Xia L, Guo Y, Zhou Q. Structural basis for the recognition of SARS-CoV-2 by full-length human ACE2. 2020 Mar 27. 367 (6485) : 1444-1448.
- Wrapp D, Wang N, Corbett KS, Goldsmith JA, Hsieh CL, Abiona O, Graham BS, McLellan JS. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. Science. 2020 Mar 13. 367 (6483): 1260-1263.
- Zhang L, Lin D, Sun X, Curth U, Drosten C, Sauerhering L, Becker S, Rox K, Hilgenfeld R. Crystal structure of SARS-CoV-2 main protease provides a basis for design of improved α-ketoamide inhibitors. 2020 Mar 20. pii : eabb3405.
- Chen YW, Yiu CB, Wong KY. Prediction of the SARS-CoV-2 (2019-nCoV) 3C-like protease (3CL (pro)) structure: virtual screening reveals velpatasvir, ledipasvir, and other drug repurposing candidates. F1000Res. 2020 Feb 21. 9:129.





